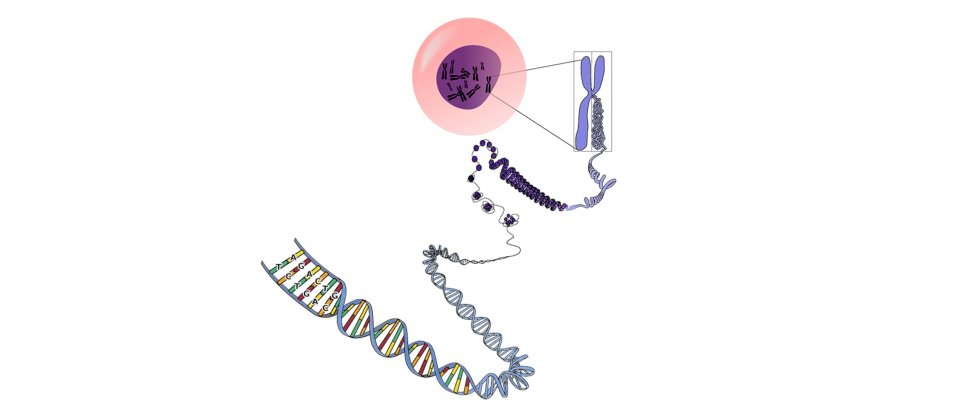
Nvidia和哈佛開發了一個深度學習工具AtacWorks,可以協助科學家進行DNA研究,即便樣本資料存在雜訊或是有所限制,在像是早期發現的癌症或是其他基因疾病研究上,都能發揮良好的作用,AtacWorks可以對定序資料進行降噪,或是辨識可存取的DNA區域,且使用Nvidia Tensor Core GPU還可縮短計算時間,將原本需要15小時才能完成的基因組預測,縮短到30分鐘以內。
人體中多數的細胞,都擁有完整的DNA,細胞核中有數十億個鹼基對,不過,每個細胞只能取用能夠發揮作用的部分,像是肝臟、血液或是皮膚細胞,不同種類的細胞,所活化的基因不同,只有決定細胞獨特功能的DNA區域會對外開放,其他區域則會被蛋白質包住,而AtacWorks則能夠讓科學家,快速地找出DNA開放區域進行研究。
AtacWorks可以與ATAC-seq搭配使用,ATAC-seq是一種在健康和生病的細胞中,尋找基因組開放區域的熱門方法,可用於探索藥物研究。不過,ATAC-seq方法有一個缺點,通常必須要使用上萬個細胞,才能取得乾淨的訊號,而這也就代表,ATAC-seq很難被用於研究,像是產生血球和血小板的幹細胞等稀有類型的細胞。
ATAC-seq方法在2013年的時候被開發出來,該方法像是一種DNA著色法,能夠對DNA開放區域上色,但是會跳過被包在蛋白質中的DNA區域,這個方法被基因組研究實驗室以及製藥公司大量採用,以檢測整個基因組中每個區域的訊號強度,了解DNA活躍的部分。當可用的細胞越少,資料出現的雜訊就越多,也就越難判斷可存取的DNA區域。
而AtacWorks是一個以PyTorch開發的卷積神經網路,其使用ATAC-seq資料集進行訓練,該模型學會了準確預測訊號的方法,研究人員發現,使用AtacWorks可以在100萬次讀取的雜訊序列中,辨識出可存取的染色質(Chromatin),相當於傳統方法需要5,000萬次讀取的乾淨資料集,如此科學家可以使用較少量的細胞進行研究,大幅降低樣品收集和定序成本。
透過將AtacWorks應用於ATAC-seq資料,現在只需要幾十個細胞,就可以獲得過去要數千數萬細胞,才能達到的實驗結果品質,因此可讓科學家更了解有關稀有類型的細胞,其活躍的DNA序列,並辨識出讓人們容易感染疾病的突變。論文共同作者哈佛大學助理教授Jason Buenrostro提到,使用AtacWorks,讓研究人員可以只用原本十分之一數量的細胞,進行單細胞實驗(Single-Cell Experiment),且GPU加速深度學習對低品質定序覆蓋進行降噪,可明顯提高稀有細胞發育和疾病相關的表觀遺傳學研究速度。
研究人員使用Tensor Core GPU進行AtacWorks分析計算,該模型花不到30分鐘,就預測出整個基因組,在具有32顆CPU的系統上,整個過程需要花費15個小時。論文的第一作者同時也是Nvidia研究人員的Avantika LalAtacWorks提到,AtacWorks不僅可以幫助降低收集染色質可存取資料的成本,對於藥物探索和診斷,也提供了新的可能性。
熱門新聞
2026-03-06
2026-03-06
2026-03-09
2026-03-09