Deepmind
Alphabet旗下專研人工智慧的DeepMind,繼兩年前以AlphaFold奪下蛋白質結構預測關鍵評估(Critical Assessment of protein Structure Prediction,CASP)所舉辦的全局距離測試(Global Distance Test,GDT)冠軍之後,今年再以AlphaFold 2創下更高的預測準確率,破解了已存在50年的生物學上的蛋白質折疊難題。
蛋白質的功能取決於它的3D結構,而其3D結構則是來自於氨基酸序列的摺疊方式。1972年的諾貝爾化學獎得主Christian Anfinsen曾提出一個假設:理論上從一個蛋白質的氨基酸序列就能判斷其結構。然而,該假設最大的挑戰在於要進入3D結構之前,蛋白質的折疊方式將是個天文數字,若要利用蠻力運算,估計有10^300種可能性,所耗費的時間可能比已知的宇宙生命還久。
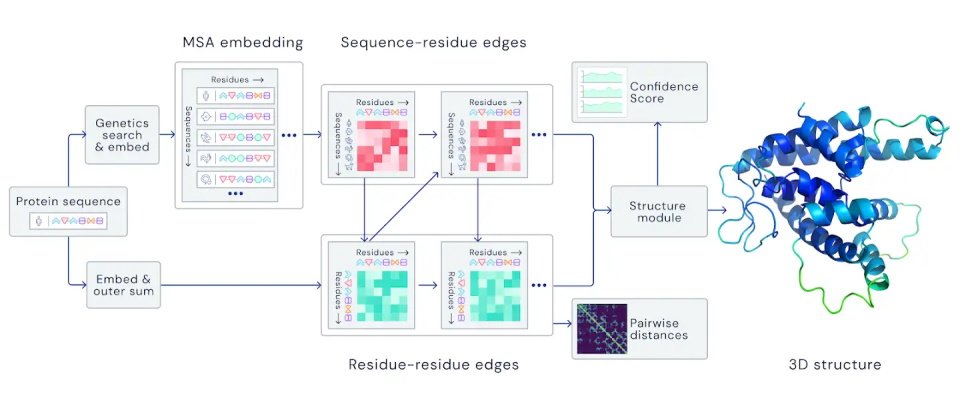
過去50年來,研究人員多是仰賴實驗技術來確定蛋白質的結構,而AlphaFold則是直接從結構著手,並不使用已知的蛋白質作為樣本,再利用兩種基於深度神經網路的方法來建構完整蛋白質結構的預測,得以預測氨基酸對之間的距離,以及連結這些氨基酸之化學鍵之間的角度。
AlphaFold是以含有17萬種蛋白質架構的蛋白質資料銀行(Protein Data Bank,PDB)數據,再加上內含未知架構之蛋白質序列的各種大型資料庫來進行訓練,以128個TPUv3核心(約等於100~200個GPU)執行數周,這樣的運算規模與現代最新大型機器學習模型差不多。
GDT則是比對各種蛋白質結構預測與已知實驗的結果,在2020年的測試中,AlphaFold 2的準確度中位數達到92.4,就算是在最難的自由建模類別的蛋白質項目中,AlphaFold 2的準確度中位數亦達87。在2018年,由AlphaFold於自由建模所創下的準確度中位數才接近60,而在AlphaFold現身以前,各種方法的準確度中位數從未超過50。
DeepMind指出,這樣的結果替生物學家開啟了以運算結構預測作為科學研究核心工具的潛力,也許對一些重要類別的蛋白質特別有用,例如因為不容易結晶而很難透過實驗來判斷的膜蛋白。
Max Planck發育生物學研究所所長Andrei Lupas則表示,AlphaFold驚人的精確度將讓他們得以解決近10年來被困住的蛋白質結構,重新啟動被擱置的專案,以了解訊號如何於細胞膜中傳輸。
DeepMind認為,更精確地判斷蛋白質架構除了能夠加速對已知疾病的了解之外,亦具備探索未知的數億種蛋白質的潛力。目前UniProt蛋白質序列資料庫存放了還在增加中的1.8億種蛋白質序列,而PDB卻只有17萬種蛋白質架構,在尚未被確認的蛋白質中,可能有一些令人興奮的新功能,而AlphaFold這類的工具則可協助科學家找到它們。
熱門新聞
2026-03-06
2026-03-06
2026-03-09
2026-03-09